Coronavirus de Wuhan
 |
| Los coronavirus se reconocen por la pequeña "corona" que tienen a su alrededor cuando se examinan en el microscopio. |
Coronavirus
Los Coronavirus son un género de virus ARN que pertenecen a la subfamilia Coronavirinae, familia Coronaviridae, del orden Nidovirales.123 Son virus envueltos con un genoma de ARN de cadena sencilla con polaridad positiva y con una nucleocápside de simetría helicoidal. El tamaño genómico de los coronavirus varía entre 26 y 31 kilobases, la más grande de un virus de ARN. El nombre coronavirus deriva de la apariencia de la envoltura bajo el microscopio electrónico de estar coronado con un anillo de estructuras redondeadas. Esta morfología está formada por proyecciones (peplómeros) de la envoltura, que son proteínas que salen de la superficie del virus y le determina el tropismo por su hospedador.
Estructura
El nombre "coronavirus" deriva del latín corona, aunque también significa halo, y hace referencia a la apariencia característica de los viriones (la forma infecciosa del virus) bajo microscopía electrónica (EM) con un margen de proyecciones grandes, superficie bulbosas que crean una que recuerda la imagen de una corona real o de la corona solar. Esta morfología es creado por los peplómeros pico viral (S), que son proteínas que pueblan la superficie del virus y determinan el tropismo de acogida. Las proteínas que contribuyen a la estructura general de todos los coronavirus son: espiculas (S), envoltura (E), la membrana (M) y de la nucleocapside (N). En el caso específico del coronavirus SARS (ver más abajo), un dominio de unión al receptor definidos en S media la unión del virus a su receptor celular, enzima convertidora de la angiotensina 2 (ACE2).1 Algunos coronavirus (específicamente los miembros de Betacoronavirus subgrupo A) también tienen un más corto pico-como proteína llamada esterasa hemaglutinina (HE).4
Las proteínas de los coronavirus que contribuyen a la estructura del virus son la proteína S, la E1 y E2 (envoltura), la M (membrana), y la proteína N (nucleocápside). En el caso específico del SARS (síndrome respiratorio agudo severo por sus siglas en inglés), un dominio de unión para receptores definidos dirigen la adherencia del virus a su receptor celular, la enzima convertidora de angiotensina 2 (ACE-2).5
Replicación
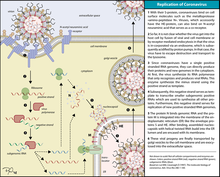
La replicación de los Coronavirus comienza con la entrada a la célula, momento en que pierde su envoltura y el genoma de ARN es depositado en el citoplasma. El genoma del coronavirus tiene un capucha metilada (CAP) en el extremo 5', y una cola poliadenilada (poly A) en el extremo 3', dándole un gran parecido al ARN mensajero del hospedador. Esto permite que el ARN se adhiera a los ribosomas para su traducción. Los coronavirus tienen también una proteína conocida como replicasa codificada en su genoma permitiendo que el ARN viral sea traducido con la maquinaria del mismo hospedador. Esta replicasa es la primera proteína que es sintetizada puesto que una vez que el gen que codifica la replicasa es traducido, el proceso se detiene por un codón de parada.
La replicación del coronavirus comienza con la entrada en la célula. A la entrada en la célula, la partícula de virus es no recubierto y el genoma de ARN se deposita en el citoplasma.
El genoma de ARN coronavirus tiene una cola 5′ metilado y una cola 3′ poliadenilado. Esto permite que el ARN se una a los ribosomas para la traducción.
Los coronavirus tienen también una proteína conocida como una replicasa codificada en su genoma que permite que el genoma viral de ARN que se transcriba en nuevas copias de ARN usando la maquinaria de la célula huésped. La replicasa es la primera proteína que al hacerse el gen se codifica la replicasa se traduce, la traducción se detiene por un codón de parada. Esto se conoce como una transcripción anidada. Cuando el transcrito de ARNm sólo se codifica en un gen, es monocistrónico. Una proteína de coronavirus no estructural proporciona una fidelidad extra para la replicación, ya que confiere una función de corrección de pruebas, que carece de enzimas de ARN polimerasa dependiente de un ARN solo.
El genoma de ARN se replica y se forma una larga poliproteína, en donde todas las proteínas están unidas. Los coronavirus tienen una proteína no estructural - una proteasa - que es capaz de separar las proteínas de la cadena. Esta es una forma de economía genética para el virus, lo que le permite codificar el mayor número de genes en un pequeño número de nucleótidos.67
En humanos
Los coronavirus fueron descritos por primera vez en la década de 1960 en las cavidades nasales de pacientes con un resfriado común. Estos virus fueron nombrados posteriormente coronavirus humano 229E y OC43 de coronavirus humano. Otros dos miembros de esta familia han sido identificados (HCoV NL63 en 2004 y HKU1 en 2005) y que han estado en infeciones mas graves del tracto respiratorio. En de enero de 2020, las muertes en China han aumentado a 17 con más de 540 casos confirmados . Se sospecha que proceden de la fauna del comercio ilegal.8
Hay siete cepas conocidas de coronavirus humanos:
- Coronavirus humano 229E (HCoV-229E)
- Coronavirus humano OC43 (HCoV-OC43)
- SARS Co-V
- Coronavirus humano NL63 (HCoV-NL63, New Haven coronavirus)
- Coronavirus humano HKU1
- Síndrome respiratorio por coronavirus de Oriente Medio (MERS-CoV), anteriormente conocido como coronavirus Novel 2012 y HCoV-CEM
- Wuhan coronavirus (2019-nCoV), también conocido como nuevo coronavirus 2019/2020 (neumonía Wuhan).910
Después de la publicación del perfil de los brotes de SARS en 2003, ha habido un renovado interés en los coronavirus entre los virólogos. Durante muchos años, los científicos sabían sobre sólo dos coronavirus humanos (HCoV-229E y OC43-HCoV). El descubrimiento de SARS-CoV añadió tercera coronavirus humano. A finales de 2004, tres laboratorios de investigación independientes informaron el descubrimiento de una cuarto coronavirus humano. Ha sido nombrado NL63, NL, y el coronavirus de New Haven por diferentes grupos de investigación. Los tres laboratorios todavía están discutiendo sobre cuál descubrió el virus de la primera y que tiene el derecho a un nombre. A principios de 2005, un equipo de investigación de la Universidad de Hong Kong informó del hallazgo de un quinto coronavirus humano en dos pacientes con neumonía. Lo llamaron HKU1 coronavirus humano. El brote de neumonía 2019-20 de China en Wuhan fue rastreado a un nuevo coronavirus, que está etiquetado como 2019-nCoV por la OMS.1112910
Síndrome respiratorio agudo severo
En 2003, tras el brote del síndrome respiratorio agudo severo (SARS), que había comenzado el año 2002 en Asia, y en el secundario en otras partes del mundo, la Organización Mundial de la Salud (OMS) emitió un comunicado de prensa indicando que un nuevo coronavirus identificado por una número de laboratorios era el agente causante del SARS. El virus fue nombrado oficialmente el coronavirus del SARS (SARS-CoV). Más de 8.000 personas fueron infectadas, alrededor del 10% de los cuales murieron.13
Síndrome respiratorio de medio oriente
En septiembre de 2012, se identificó un nuevo tipo de coronavirus, llamado inicialmente nuevo coronavirus 2012, y ahora el nombre oficial de Oriente Medio síndrome respiratorio coronavirus (MERS-CoV). La Organización Mundial de la Salud emitió una alerta mundial pronto después. La actualización de la OMS el 28 de septiembre 2012 declaró que no parecía el virus de transmitirse fácilmente de persona a persona. Sin embargo, el 12 de mayo de 2013, un caso de transmisión de humano a humano en Francia fue confirmada por el Ministerio de Asuntos Sociales y Salud de Francia. Además, los casos de transmisión de humano a humano han sido reportados por el Ministerio de Salud de Túnez. Dos casos confirmados parecen haber contraído la enfermedad de su difunto padre, que se enfermaron después de una visita a Qatar y Arabia Saudita. A pesar de esto, parece que el virus tiene problemas para la difusión de humano a humano, como la mayoría de las personas que están infectadas no transmiten el virus.141516171819
Para el 30 de octubre de 2013, había 124 casos y 52 muertes en Arabia Saudita. Después de que el Centro Médico Erasmus holandesa secuenciado el virus, el virus se le dio un nuevo nombre, Human coronavirus-Centro Médico Erasmus (HCoV-CEM). El nombre final para el virus es Oriente Medio síndrome respiratorio coronavirus (MERS-CoV). En el 2014 de mayo, los dos únicos casos de infección Estados Unidos MERS-CoV se registraron, tanto que ocurren en trabajadores de la salud que trabajaban en Arabia Saudita y luego se desplazaron a Estados Unidos. Ambos de estos individuos fueron hospitalizados temporalmente y luego dados de alta. En mayo de 2015, un brote de MERS-CoV se produjo en Corea del Sur, cuando un hombre que había viajado a Oriente Medio, visitó 4 hospitales diferentes en el área de Seúl para tratar su enfermedad. Esto provocó uno de los mayores brotes de MERS-CoV fuera del Medio Oriente. En diciembre de 2019, 2.468 casos de infección MERS-CoV habían sido confirmados por pruebas de laboratorio, de los cuales 851 fueron mortales, una tasa de mortalidad de aproximadamente el 34,5%.20212223
En la medicina veterinaria
Los coronavirus han sido reconocidos como causantes de condiciones patológicas en las veterinaria desde principios de 1970. A excepción de infecciosa aviar bronquitis, las principales enfermedades relacionadas tienen principalmente una intestinal ubicación.
Los coronavirus infectan principalmente el tracto respiratorio y gastrointestinal superior de mamíferos y aves. Actualmente hay siete cepas conocidas del coronavirus que infecta a los humanos. Los coronavirus se cree que causa un porcentaje significativo de todos los resfriados comunes en adultos y niños humanos. Los coronavirus causan los resfriados con síntomas importantes, por ejemplo, fiebre, inflamación de las adenoides de la garganta, en los seres humanos principalmente en las temporadas de invierno y primavera temprana. Los coronavirus puede causar neumonía, ya sea directa neumonía viral o una neumonía bacteriana secundaria, y la bronquitis, ya sea bronquitis viral directa o una bronquitis bacteriana secundaria. El coronavirus humano muy conocido fue descubierto en 2003, SARS-CoV que causa el síndrome respiratorio agudo severo (SARS), tiene una patogénesis único porque hace que tanto superior e inferior infecciones de las vías respiratorias. La importancia económica y el impacto de los coronavirus como agentes causantes del resfriado común son difíciles de evaluar debido a que, a diferencia de los rinovirus (otro virus del resfriado común), coronavirus humanos son difíciles de cultivar en el laboratorio.2425
Los coronavirus también causa una serie de enfermedades en los animales de granja y mascotas domesticadas, algunos de los cuales pueden ser graves y son una amenaza para la industria agrícola. En los pollos, el virus de la bronquitis infecciosa (IBV), un coronavirus, se dirige no sólo las vías respiratorias, sino también el tracto urogenital. El virus puede propagarse a los diferentes órganos a través de los coronavirus de la gallina. Con consecuencias economicamente significativas en los animales de granja incluyen coronavirus porcino (coronavirus de la gastroenteritis transmisible, TGE) y coronavirus bovino, que da resultado de diarrea en los animales jóvenes. Coronavirus felino: existen dos formas, coronavirus entérico felino es un patógeno de importancia clínica menor, pero la mutación espontánea de este virus puede provocar una peritonitis infecciosa felina (FIP), una enfermedad asociada con una alta mortalidad. Del mismo modo, hay dos tipos de coronavirus que infectan a los hurones: hurón entérico coronavirus causa un síndrome gastrointestinal conocido como epizoótica catarral enteritis (ECE), y una versión sistémica más letal del virus (como FIP en los gatos), conocido en hurones como coronavirus sistémica hurón (FSC). Hay dos tipos de coronavirus canino (CoVC), uno que causa la enfermedad gastrointestinal leve y uno que se ha encontrado que causa enfermedad respiratoria. El virus de la hepatitis del ratón (MHV) es un coronavirus que causa una enfermedad epidémica murino con una alta mortalidad, especialmente entre las colonias de ratones de laboratorio.262728
El coronavirus HKU2 esta relacionada con diarrea aguda porcina síndrome de coronavirus (SADS-CoV) causa diarrea en cerdos.29
Antes del descubrimiento de SARS-CoV, MHV había sido estudiado coronavirus tanto in vivo como in vitro, así como a el nivel molecular. Algunas cepas de MHV antes de causar una desmielinizante progresiva encefalitis en ratones a los que se ha utilizado como un modelo murino para la esclerosis múltiple. Importantes esfuerzos de investigación se han centrado en dilucidar la patogénesis viral de estos coronavirus animales, especialmente por los virólogos interesados en las enfermedades zoonóticas y veterinarios.30
(Que aparece después de su importancia económica estimada)
- Virus de la bronquitis infecciosa (IBV) causas infecciosa aviar bronquitis.
- Porcino coronavirus (transmisible gastroenteritis coronavirus de cerdos, TGEV).
- Coronavirus bovino (BCV), responsable de la profusa severa enteritis en los terneros jóvenes.
- Coronavirus felino (FCoV) provoca enteritis leve en los gatos, así como graves peritonitis infecciosa felina (otras variantes del mismo virus).
- Los dos tipos de canino coronavirus (CoVC) (una enteritis causando, la otra que se encuentra en las enfermedades respiratorias).
- Coronavirus turco (TCV) provoca enteritis en pavos.
- Ferret entérica coronavirus provoca enteritis catarral epizoótica en hurón s.
- Ferret sistémica coronavirus causa FIP-como el síndrome sistémico en hurones.
- Pantrópicas coronavirus canino.
- Coronavirus Canino
Otra nueva enfermedad veterinaria, porcina virus de la diarrea epidémica (PED o PEDV), ha surgido en todo el mundo. Su importancia económica es todavía poco clara, pero muestra una alta mortalidad en los lechones.323334
Historia Natural
Evolución
El ancestro común más reciente del coronavirus se ha colocado a 8000 antes de Cristo. Ellos pueden ser considerablemente mayores que esto. Otra estimación coloca el ancestro común más reciente (MRCA) de todos los coronavirus alrededor de 8100 aC. El ancestro común más reciente de Alphacoronavirus, Betacoronavirus, Gammacoronavirus y Deltacoronavirus se han datado en alrededor de 2400 AC, 3300 AC, 2800 AC y 3000 AC, respectivamente. Al parecer los murciélagos y pájaros, la sangre caliente vertebrados voladores, son anfitriones ideales para la fuente de genes de coronavirus. El Coronavirus bovino y canino respiratorio con el coronavirus divergieron de un ancestro común en 1951. El coronavirus bovino y coronavirus humano OC43 se separaron en 1899. El coronavirus bovino se separaron de la especie equina coronavirus al final del siglo 18. Otra estimación sugiere que coronavirus humano OC43 divergió de coronavirus bovino en 1890. El ancestro común más reciente de coronavirus humano OC43 ha sido fechado en la década de 1950. El síndrome respiratorio Oriente Medio coronavirus, aunque relacionado con varias especies de murciélagos parece haber divergido de estos hace varios siglos. El coronavirus de murcielago esán más estrechamente relacionados y el coronavirus del SARS que se separaron en 1986.3536373839404142434445
Taxonomía
- Género 'Betacoronavirus'; especie tipo: murino coronavirus
- Especie: Betacoronavirus 1, Human coronavirus HKU1, murino coronavirus, Pipistrellus Bat coronavirus HKU5, Rousettus Bat coronavirus HKU9, agudo severo síndrome respiratorio relacionado con coronavirus, tylonycteris Bat coronavirus HKU4, MERS-CoV, coronavirus humano OC43, Hedgehog coronavirus (EriCoV), Wuhan coronavirus (2019-nCoV)
- Género 'Gammacoronavirus'; especie tipo: aviar coronavirus
- Género 'Deltacoronavirus'; especie tipo: Bulbul coronavirus HKU11
Otras cepas
- IBV Aviar
- Coronavirus Entérico Humano
- Virus de la Hepatitis Murina
- Coronavirus de la Diarrea Neonatal del Becerro
- Virus de la Encefalitis Hemaglutinante Porcina
- Virus de la Gastroenteritis Transmisible Porcina
- Coronavirus de Ratas
- Virus Sialodacrioadenitis
Referencias
- ↑a b AMQ King; E Lefkowitz; MJ Adams et al., eds. (2011). «Family Coronaviridae». Ninth Report of the International Committee on Taxonomy of Viruses. Elsevier, Oxford. pp. 806-828. ISBN 978-0-12-384684-6. Parámetro desconocido
|vauthors=ignorado (ayuda); - ↑ International Committee on Taxonomy of Viruses (24 August 2010). «ICTV Master Species List 2009 – v10» (xls).
- ↑ Thiel V (editor). (2007). Coronaviruses: Molecular and Cellular Biology (1st ed. edición). Caister Academic Press. ISBN 978-1-904455-16-5 .
- ↑ «Structure of SARS coronavirus spike receptor-binding domain complexed with receptor». Science 309 (5742): 1864-1868. September 2005. Bibcode:2005Sci...309.1864L. PMID 16166518. doi:10.1126/science.1116480. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ Li, Fang, et. al. (2005). «Structure of SARS Coronavirus Spike Receptor-Binding Domain Complexed with Receptor». Science 309: 1864-1868.
- ↑ «Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens». Journal of Virology 90 (16): 7415-7428. August 2016. PMC 4984655. PMID 27279608. doi:10.1128/JVI.00080-16. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Coronaviruses: an overview of their replication and pathogenesis». Methods in Molecular Biology 1282. 2015. pp. 1-23. ISBN 978-1-4939-2437-0. PMC 4369385. PMID 25720466. doi:10.1007/978-1-4939-2438-7_1. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Human coronaviruses: insights into environmental resistance and its influence on the development of new antiseptic strategies». Viruses 4 (11): 3044-3068. November 2012. PMC 3509683. PMID 23202515. doi:10.3390/v4113044. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑a b «Laboratory testing of human suspected cases of novel coronavirus (nCoV) infection. Interim guidance, 10 January 2020». Archivado desde el original el 20 January 2020. Consultado el 14 January 2020. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑a b «Novel Coronavirus 2019 (nCoV-2019), Wuhan, China». Archivado desde el original el 14 January 2020. Consultado el 14 January 2020. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ «Identification of a new human coronavirus». Nature Medicine 10 (4): 368-373. April 2004. PMID 15034574. doi:10.1038/nm1024. Archivado desde el original el 21 August 2007. Consultado el 12 September 2019. Parámetro desconocido
|vauthors=ignorado (ayuda); Parámetro desconocido|url-status=ignorado (ayuda) - ↑ «WHO Statement Regarding Cluster of Pneumonia Cases in Wuhan, China». www.who.int (en inglés). 9 January 2020. Archivado desde el original el 14 January 2020. Consultado el 10 January 2020. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ «Structure of SARS coronavirus spike receptor-binding domain complexed with receptor». Science 309 (5742): 1864-1868. September 2005. Bibcode:2005Sci...309.1864L. PMID 16166518. doi:10.1126/science.1116480. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ CDC (2 August 2019). «MERS Transmission». Centers for Disease Control and Prevention (en inglés estadounidense). Archivado desde el original el 7 December 2019. Consultado el 10 December 2019. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ Nouveau coronavirus – Point de situation : Un nouveau cas d’infection confirmé (enlace roto disponible en este archivo). (Novel coronavirus – Status report: A new case of confirmed infection) 12 May 2013 social-sante.gouv.fr
- ↑ Doucleef, Michaeleen (26 September 2012). «Scientists Go Deep On Genes Of SARS-Like Virus». Associated Press. Archivado desde el original el 27 September 2012. Consultado el 27 September 2012. Parámetro desconocido
|name-list-format=ignorado (ayuda); Parámetro desconocido|url-status=ignorado (ayuda) - ↑ Falco, Miriam (24 September 2012). «New SARS-like virus poses medical mystery». CNN Health. Archivado desde el original el 1 November 2013. Consultado el 16 March 2013. Parámetro desconocido
|name-list-format=ignorado (ayuda); Parámetro desconocido|url-status=ignorado (ayuda) - ↑ «New SARS-like virus found in Middle East». Al-Jazeera. 24 September 2012. Archivado desde el original el 9 March 2013. Consultado el 16 March 2013. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ Kelland, Kate (28 September 2012). «New virus not spreading easily between people: WHO». Reuters. Archivado desde el original el 24 November 2012. Consultado el 16 March 2013. Parámetro desconocido
|name-list-format=ignorado (ayuda); Parámetro desconocido|url-status=ignorado (ayuda) - ↑ «Novel coronavirus infection - update». World Health Association. 22 May 2013. Archivado desde el original el 7 June 2013. Consultado el 23 May 2013. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ CDC (2 August 2019). «MERS in the U.S.». Centers for Disease Control and Prevention (en inglés estadounidense). Archivado desde el original el 15 December 2019. Consultado el 10 December 2019. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ Sang-Hun, Choe (8 June 2015). «MERS Virus's Path: One Man, Many South Korean Hospitals». The New York Times. Archivado desde el original el 15 July 2017. Consultado el 1 March 2017. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ «Middle East respiratory syndrome coronavirus (MERS-CoV)». WHO. Archivado desde el original el 18 October 2019. Consultado el 10 December 2019. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ «Prevalence and genetic diversity analysis of human coronaviruses among cross-border children». Virology Journal (en inglés) 14 (1): 230. November 2017. PMC 5700739. PMID 29166910. doi:10.1186/s12985-017-0896-0. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Healthcare-associated atypical pneumonia». Seminars in Respiratory and Critical Care Medicine 30 (1): 67-85. February 2009. PMID 19199189. doi:10.1055/s-0028-1119811. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ Murray, Jerry (16 April 2014). «What's New With Ferret FIP-like Disease?» (xls). Archivado desde el original el 24 April 2014. Consultado el 24 April 2014. Parámetro desconocido
|name-list-format=ignorado (ayuda); Parámetro desconocido|url-status=ignorado (ayuda) - ↑ «Progress and challenges toward the development of vaccines against avian infectious bronchitis». Journal of Immunology Research 2015: 1-12. 2015. PMC 4411447. PMID 25954763. doi:10.1155/2015/424860. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Coronavirus pathogenesis and the emerging pathogen severe acute respiratory syndrome coronavirus». Microbiology and Molecular Biology Reviews 69 (4): 635-664. December 2005. PMC 1306801. PMID 16339739. doi:10.1128/MMBR.69.4.635-664.2005. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin (enlace roto disponible en este archivo)., Peng Zhou, Hang Fan, Tian Lan, Xing-Lou Yang, Wei-Feng Shi, Wei Zhang, Yan Zhu, Ya-Wei Zhang, Qing-Mei Xie, Shailendra Mani, Xiao-Shuang Zheng, Bei Li, Jin-Man Li, Hua Guo, Guang-Qian Pei, Xiao-Ping An, Jun-Wei Chen, Ling Zhou, Kai-Jie Mai, Zi-Xian Wu, Di Li, Danielle E. Anderson, Li-Biao Zhang, Shi-Yue Li, Zhi-Qiang Mi, Tong-Tong He, Feng Cong, Peng-Ju Guo, Ren Huang, Yun Luo, Xiang-Ling Liu, Jing Chen, Yong Huang, Qiang Sun, Xiang-Li-Lan Zhang, Yuan-Yuan Wang, Shao-Zhen Xing, Yan-Shan Chen, Yuan Sun, Juan Li, Peter Daszak, Lin-Fa Wang, Zheng-Li Shi, Yi-Gang Tong & Jing-Yun Ma, Nature, 5 April 2018.
- ↑ «Cell replacement therapies to promote remyelination in a viral model of demyelination». Journal of Neuroimmunology 224 (1–2): 101-107. July 2010. PMC 2919340. PMID 20627412. doi:10.1016/j.jneuroim.2010.05.013. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens». Journal of Virology 90 (16): 7415-7428. August 2016. PMC 4984655. PMID 27279608. doi:10.1128/JVI.00080-16. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Coronavirus gene 7 counteracts host defenses and modulates virus virulence». PLoS Pathogens 7 (6): e1002090. June 2011. PMC 3111541. PMID 21695242. doi:10.1371/journal.ppat.1002090. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Alphacoronavirus protein 7 modulates host innate immune response». Journal of Virology 87 (17): 9754-67. September 2013. PMC 3754097. PMID 23824792. doi:10.1128/JVI.01032-13. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «the Merck Veterinary Manual». Archivado desde el original el 9 October 2013. Consultado el 1 June 2013. Parámetro desconocido
|url-status=ignorado (ayuda) - ↑ «Discovery of seven novel Mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavirus». Journal of Virology 86 (7): 3995-4008. April 2012. PMC 3302495. PMID 22278237. doi:10.1128/JVI.06540-11. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Evolutionary dynamics of bovine coronaviruses: natural selection pattern of the spike gene implies adaptive evolution of the strains». The Journal of General Virology 94 (Pt 9): 2036-2049. September 2013. PMID 23804565. doi:10.1099/vir.0.054940-0. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Complete genomic sequence of human coronavirus OC43: molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event». Journal of Virology 79 (3): 1595-1604. February 2005. PMC 544107. PMID 15650185. doi:10.1128/jvi.79.3.1595-1604.2005. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Genetic characterization of Betacoronavirus lineage C viruses in bats reveals marked sequence divergence in the spike protein of pipistrellus bat coronavirus HKU5 in Japanese pipistrelle: implications for the origin of the novel Middle East respiratory syndrome coronavirus». Journal of Virology 87 (15): 8638-8650. August 2013. PMC 3719811. PMID 23720729. doi:10.1128/JVI.01055-13. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Molecular epidemiology of human coronavirus OC43 reveals evolution of different genotypes over time and recent emergence of a novel genotype due to natural recombination». Journal of Virology 85 (21): 11325-11337. November 2011. PMC 3194943. PMID 21849456. doi:10.1128/JVI.05512-11. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «A case for the ancient origin of coronaviruses». Journal of Virology 87 (12): 7039-7045. June 2013. PMC 3676139. PMID 23596293. doi:10.1128/JVI.03273-12. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Evolutionary insights into the ecology of coronaviruses». Journal of Virology 81 (8): 4012-4020. April 2007. PMC 1866124. PMID 17267506. doi:10.1128/jvi.02605-06. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «SARS-Coronavirus ancestor's foot-prints in South-East Asian bat colonies and the refuge theory». Infection, Genetics and Evolution 11 (7): 1690-702. October 2011. PMID 21763784. doi:10.1016/j.meegid.2011.06.021. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Evolutionary relationships between bat coronaviruses and their hosts». Emerging Infectious Diseases 13 (10): 1526-1532. October 2007. PMC 2851503. PMID 18258002. doi:10.3201/eid1310.070448. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Identification and characterization of a novel alpaca respiratory coronavirus most closely related to the human coronavirus 229E». Viruses 4 (12): 3689-3700. December 2012. PMC 3528286. PMID 23235471. doi:10.3390/v4123689. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Evidence supporting a zoonotic origin of human coronavirus strain NL63». Journal of Virology 86 (23): 12816-12825. December 2012. PMC 3497669. PMID 22993147. doi:10.1128/JVI.00906-12. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Coronavirus gene 7 counteracts host defenses and modulates virus virulence». PLoS Pathogens 7 (6): e1002090. June 2011. PMC 3111541. PMID 21695242. doi:10.1371/journal.ppat.1002090. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ «Alphacoronavirus protein 7 modulates host innate immune response». Journal of Virology 87 (17): 9754-67. September 2013. PMC 3754097. PMID 23824792. doi:10.1128/JVI.01032-13. Parámetro desconocido
|vauthors=ignorado (ayuda)




Comentarios
Publicar un comentario